|
用于悬浮/贴壁哺乳动物真核细胞的大容量核酸转染仪

LONZA 4D-Nucleofector LV 大规模细胞核转染系统(4D-Nucleofector LV Unit,简称LV模块,或LV单元),是在传统电穿孔技术的基础上,针对每一种细胞,优化电脉冲参数、转染液,使细胞转染效率、细胞活率得到显著提升,解决了原代细胞、干细胞、T细胞等使用传统电穿孔转染效率低下的问题。
4D-Nucleofector核转技术(原德国Amaxa Nucleofection核转染技术)对质粒DNA转染效率高达90%,寡核苷酸(如siRNA)转染效率高达99%,面世20年来,已成功转染>1200种细胞系、>130种原代细胞,并在干细胞、免疫细胞、神经细胞等各类较难转染的细胞中获得了优秀的转染效果。
同时,4D-Nucleofector核转染可让外源基因直接穿过核膜进入细胞核,不依赖细胞有丝,实现2小时快速表达。LONZA 4D-Nucleofector LV模块可提供单次最高2×10^9个细胞、封闭、无菌的高效转染,在人T细胞、CHO悬浮细胞、HEK23悬浮细胞、K562细胞等超过700种细胞中大量应用。
LONZA全球共享数据库,包含超过1500种原代细胞、细胞系的转染数据,提供包括细胞来源、传代、培养条件、培养基、到转染后培养技巧的成熟流程,让研究者在海量数据支持中快速开展实验,并取得成功。操作4D-Nucleofector核转染设备时,只需一键调用LONZA内置的转染程序,即可实现细胞快速、高效转染,而无需自行摸索转染条件。
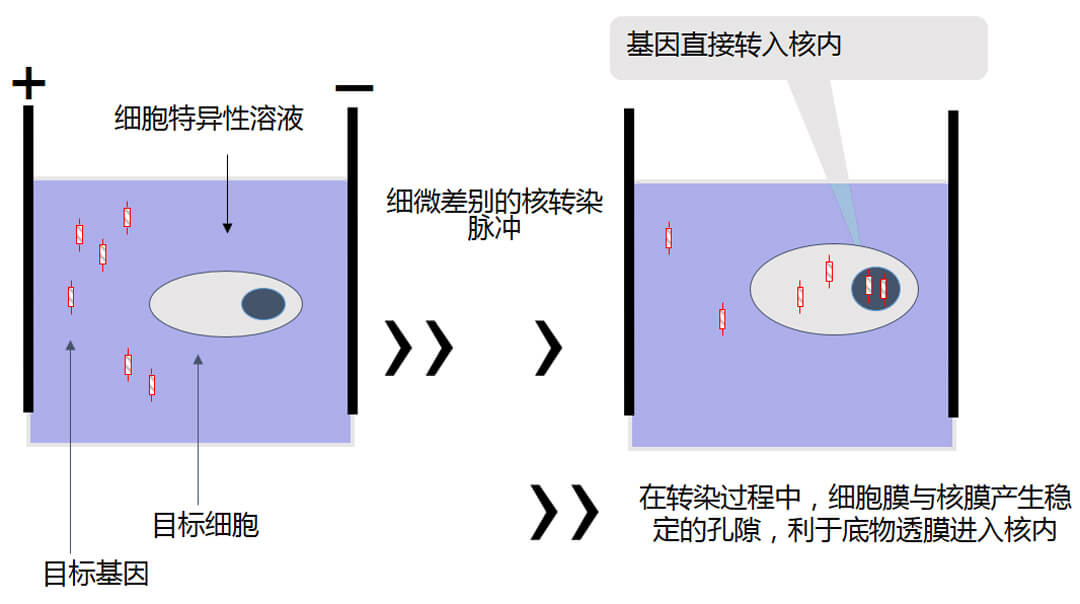
4D-Nucleofector核转染技术优势
-采用高分子聚合物电极,避免传统铝电极的离子毒害,维持细胞生理状态,提高存活率。
-转染后可快速观察实验结果,例如转染GFP后最快2小时可观察到蛋白。
-无需自己优化条件,拥有超过650种细胞类型的现成程序,可直接使用,全球共享数据库,超过1500条转染数据可供参考。
-可转染包括DNA、mRNA、miRNA、siRNA、肽、蛋白质在内的多种底物。
-可用于难以转染的原代细胞、干细胞、神经元、细胞系。
-灵活的转染规模缩放,可在低、中、高通量中轻松转移,实现2×10^4至1×10^9个细胞数量的轻松扩展。
-已在全球8000多家同行评议的出版物中发表。
LONZA 4D-Nucleofector LV大规模细胞核转染仪,细胞治疗基础研究高效电转染系统
1、封闭、无菌的系统和耗材设计,满足细胞治疗基础研究的需求。

2、20mL大容量,LV核转仪提供单次1mL(最多1×10^8个细胞)的手动加样、20×1mL(最多2×10^9个细胞)的自动加样,快速高效。

3、符合cGMP要求,可使用符合美国FDA21 CFR part 11、欧盟Annex 11的软件进行控制,适用于严格监管的GMP实验室。
①用户管理(登陆,不同级别)
②具有用户姓名与时间标记的电子签名
③任何记录的修改或是数据的创建均被记录在案
④结果报告:失败报告,以及详细的问题描述
⑤可根据21CFR part11的要求导出数据(加密,预防)
⑥审计追踪(使用记录、更改记录)
⑦无数据删除可能
4、规模可简单放大,在4D-Nucleofector X模块(4D-Nucleofector X Unit,简称X模块,或X单元)上进行的2×10^4细胞数量的优化转染方案,可直接在LV模块上使用。

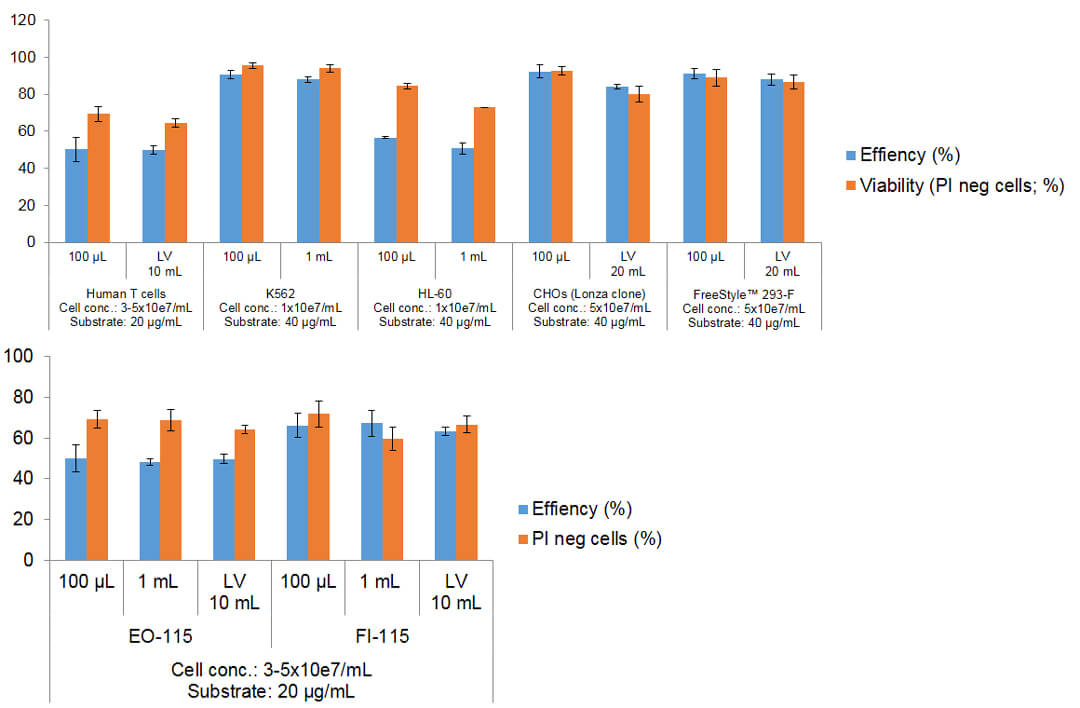
实验数据:分别用4D-Nucleofector细胞核转仪X模块(100μL)、LV模块(1mL)向包括人T细胞、PBMC、293细胞、CHO细胞在内多种代表性细胞中转染pmaxGFP载体,实现转染规模的线性放大。
参考文献
1.Closed-system transposon-mediated manufacture of GMP grade CAR T-cells via the Lonza Nucleofector LV XL. LM Brownrigg,et al. ELSERIER(2020)
2.Biomimetic mineralized collagen scaffolds and their effect on osteogenic differentiation. L Luo,et al. ELSERIER(2020)
3.Optimization of clarification process for viral vectors. B Raghavan,et al. ELSERIER(2020)
4.Generation and testing of clinical-grade exosomes for pancreatic cancer. Mayela Mendt,et al. JCI Insight(2018)
5.Off the shelf T cell therapies for hematologic malignancies. BJ McCreedy,et al. ELSERIER(2018)
6.Unlocking the Potential of CRISPR-Based Gene Editing. Andrea Toell. Genetic Engineering & Biotechnology News(2017)
|